ニュース&イベント NEWS & EVENTS
L-アミノ酸を選択する反応メカニズムの解明、生命進化の大きな謎の解明に一歩前進
~量子力学と分子力学のハイブリッド計算法によるRNAのキラル選択的アミノアシル化反応の可視化~
研究の要旨とポイント
- 地球上の生物はなぜL-アミノ酸(左手型アミノ酸)を利用するようになったのかという「アミノ酸のホモキラリティー」の起源問題は、生命進化の大きな謎のひとつであり、その鍵を握るものとして「L-アミノ酸選択的なRNAのアミノアシル化反応」が注目されています。
- シュレディンガー方程式に基づいた量子力学計算とニュートンの古典力学に基づいた分子力学・分子動力学計算を組み合わせたシミュレーション法によって、遷移状態を含むL-アミノ酸選択的なRNAのアミノアシル化反応全体のメカニズムを初めて明らかにしました。
- アミノ酸のホモキラリティーを考える上で重要な知見であると同時に、キラル選択的な反応を制御するタンパク質工学・核酸工学への新たな展開も期待できます。
東京理科大学先進工学部電子システム工学科の安藤格士准教授、同学部生命システム工学科の田村浩二教授は、シュレディンガー方程式に基づいた量子力学計算とニュートンの古典力学に基づいた分子力学・分子動力学計算を組み合わせたシミュレーション法によって、遷移状態を含む「L-アミノ酸選択的なRNAのアミノアシル化反応」の全体のメカニズムを初めて明らかにし、実験においては不明であったL-アミノ酸が選択的に反応しやすい理由を計算機シミュレーションで解明しました。
地球上の生物はなぜL-アミノ酸(左手型アミノ酸)を利用するようになったのかという問いは、「アミノ酸のホモキラリティー」の起源問題と呼ばれ、生命進化の大きな謎のひとつでした。「L-アミノ酸選択的なRNAのアミノアシル化反応」は、この問題を解決する重要な反応であると考えられていますが、この反応において、なぜL-アミノ酸が選ばれるのかについては、全くわかっていませんでした。今回、研究チームは、この詳細なメカニズムに関して、シミュレーションに量子力学計算を取り入れることにより、「キラル選択的アミノアシル化反応」における遷移状態を含む反応全体のメカニズムを初めて明らかにしました。
この結果は長年謎であった「タンパク質はなぜL-アミノ酸から構成されているのか?」という問題解決に重要な光を投げかけると同時に、キラル選択的な反応を制御するタンパク質工学・核酸工学への新たな展開も期待できます。
本研究成果は国際雑誌『Life』に3月7日付けでオンライン掲載されました。
研究の背景
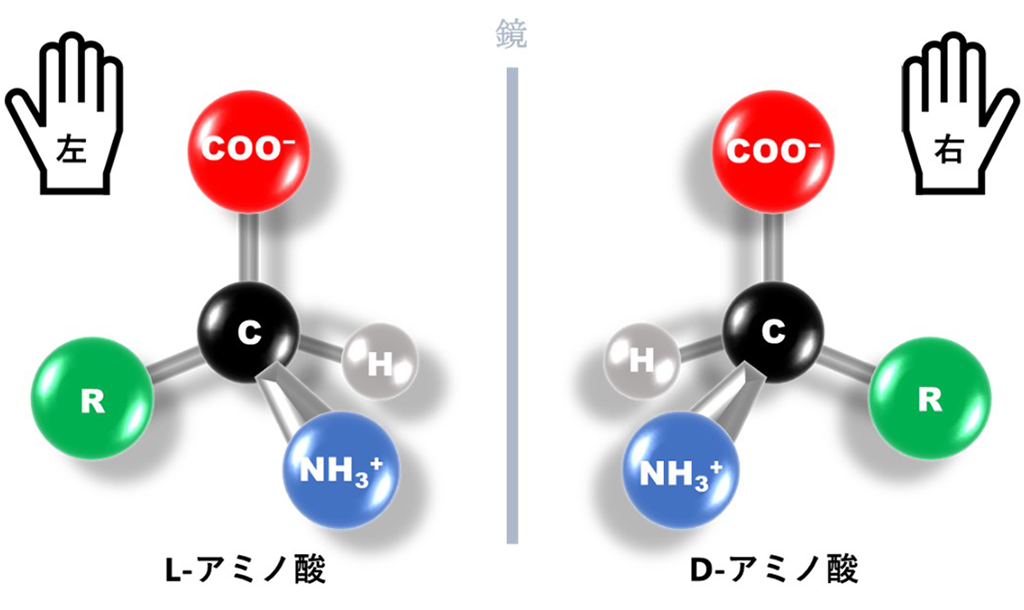
地球上の生命は、分子レベルでは極めて非対称性を持ったものとして存在しており、核酸はD型(右手型)の糖から、タンパク質はL型(左手型)のアミノ酸(*1)からのみ構成されています(図1)。タンパク質は、アミノ酸が鎖状につながった分子であり、宇宙には、L型とD型という鏡像関係の構造をもつアミノ酸がほぼ同量で存在しているにもかかわらず、現在の生物は、鏡像異性体をもたないグリシンを除いて、L-アミノ酸のみを利用してタンパク質を合成しています。アミノ酸が一方のキラリティーからのみ構成されていることは「アミノ酸のホモキラリティー」と呼ばれていますが、その起源については様々な説が唱えられてきました。
素粒子が持つ本質的な対称性の破れの結果として、L-アミノ酸がD-アミノ酸よりもごくわずかに多く存在し、宇宙空間からL-アミノ酸が彗星などによって原始地球に運ばれた結果として、L-アミノ酸から構成される原始生命体ができたという考えがあります。しかしながら、隕石中にわずかに見られる過剰なL-アミノ酸は、α炭素がメチル化されたアミノ酸であり、容易にラセミ化する(L型とD型が等量混在する状態になる)通常のα-アミノ酸(タンパク質の構成成分)は、L型とD型がほぼ等量で見つかっています。仮にL-アミノ酸が原始地球にわずかに過剰に存在したとしても、それがL-アミノ酸のみから構成されるタンパク質を形成するまでには、ラセミ化の壁を乗り越える必要があり、これが、即、「アミノ酸のホモキラリティー」の起源につながるとは言い難いと考えられます。
アミノ酸のホモキラリティーの起源を考える上においては、生命の起源と進化の連続性を考える必要があります。現在の地球上の生物では、タンパク質を合成する最初のステップで、アミノアシルtRNA合成酵素という酵素が、対応するL-アミノ酸とそのアミノ酸の運搬を担うtRNA(*2)を厳密に認識し、互いを結合させています。田村教授らは、tRNAのアミノアシル化反応に注目し、2004年に、この反応を模倣した原始モデル系で、アミノアシルtRNA合成酵素がない状態でも、L-アミノ酸が優先的にRNAに結合される現象(「キラル選択的アミノアシル化反応(*3)」)を発見しました(Tamura and Schimmel, Science, 305, 1253 (2004))。現存生物の誕生前の地球には、RNAが遺伝情報を保持するとともに化学反応も触媒する「RNAワールド」が存在したとの仮説があり、田村教授らの発見は、アミノ酸のホモキラリティーはRNAワールドにおけるtRNAのアミノアシル化反応を起源にしていることを強く示唆しています。
しかしながら、この反応において、なぜL-アミノ酸が選ばれるのかについては、まったく不明でした。安藤准教授と田村教授は、この反応で、なぜL-アミノ酸が選ばれるのかについての詳細なメカニズムに関して、古典力学に準じて分子のエネルギーを計算する分子力学計算(*4)、および分子の動きを原子のレベルでシミュレーションすることができる分子動力学法(*5)を用いて解析を進めてきました(Ando et al., Nucleic Acid Res., 46, 11144 (2018))。今回、シミュレーションに量子力学計算(*6)を取り入れることにより、これまでには知ることのできなかった、「キラル選択的アミノアシル化反応」における遷移状態を含む反応全体のメカニズムを初めて明らかにしました。
研究成果の概要
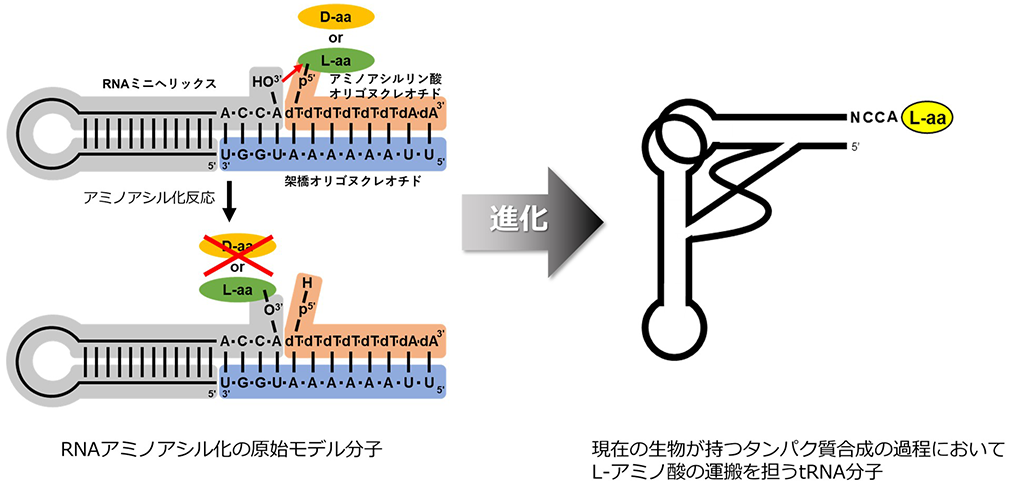
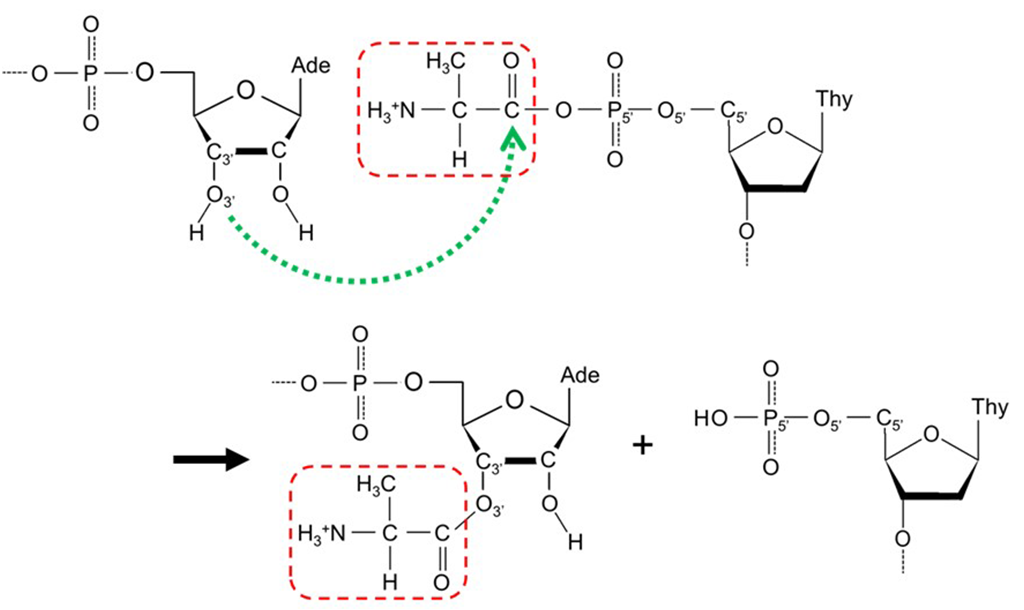
田村教授らが2004年の実験研究で用いたモデル分子系は、tRNAの原始型と考えられる「RNAミニヘリックス」に加え、さらに「アミノアシルリン酸オリゴヌクレオチド」と「架橋オリゴヌクレオチド」から構成されています(図2)。アミノアシル化反応は、RNAミニヘリックスの3’位のヒドロキシル基(-OH)がアミノアシルリン酸オリゴヌクレオチドのカルボニル炭素(-C(=O)-)を求核攻撃することで開始されます(図3)。このモデル反応系で使われるアミノアシルリン酸オリゴヌクレオチドは、非常に不安定な化合物であり、X線回折などの手法を用いての構造解析が困難なため、そのメカニズムの詳細は長く不明でした。
RNAミニヘリックスのキラル選択的アミノアシル化反応が報告されてから14年後の2018年に、安藤准教授と田村教授らは、反応部位を含むRNA分子の分子動力学(Molecular Dynamics: MD)シミュレーションを用いて、この求核反応中で許される原子間の距離、原子が近づく角度を、化学的、および、幾何学的観点から詳細に解析し、L-アミノ酸の選択性を決定するメカニズムを議論しました。一般的に原子間の結合の解離、生成を伴う化学反応のメカニズムを知るためには、遷移状態を含む、分子の反応前の状態から反応後の状態へと移り変わる様子を調べることが必須となっています。しかしながら、分子の立体構造のエネルギーを経験的な関数で計算する分子力学(Molecular Mechanics: MM)計算、および、そのエネルギー計算を基にニュートンの運動方程式に準じて原子・分子を運動させるMD法では、化学反応を直接的にシミュレーションすることができません。これがキラル選択的なアミノアシル化反応の詳細なメカニズムを議論する上で大きな問題として残されていました。
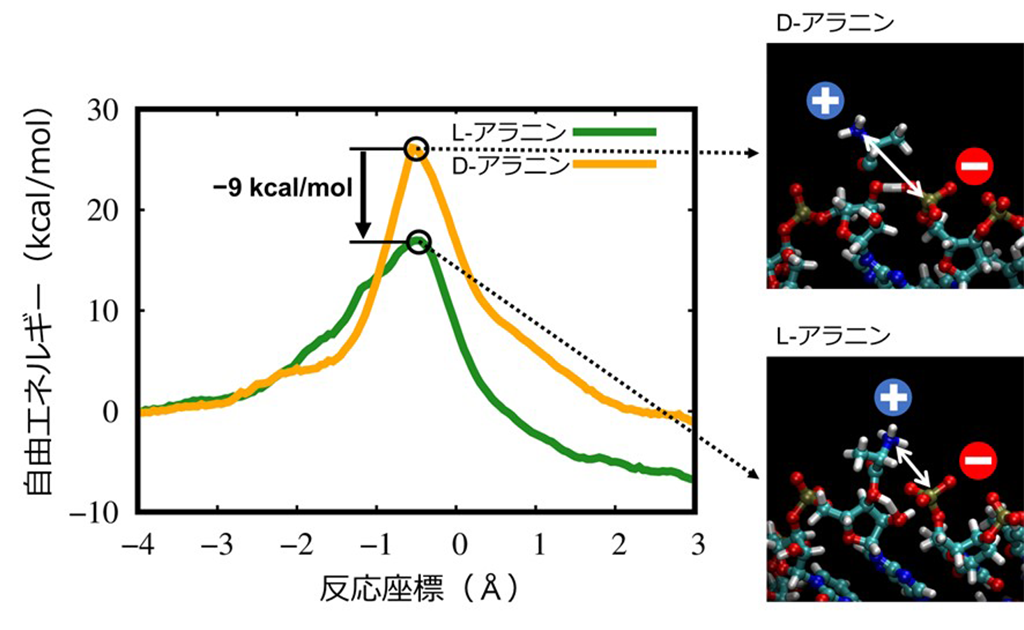
今回の研究では、化学反応を扱うことが可能な量子力学(Quantum Mechanics: QM)計算と従来の分子力学・分子動力学計算を組み合わせたQM/MM MD法(*7)を用いることで残されていた問題を解決し、RNAのアミノアシル化の全反応過程をシミュレートすることに成功しました。また、反応自由エネルギー変化の効率な計算を可能とするアンブレラサンプリング法(*8)をQM/MM MD法に組み合わせることにより、L-アラニンとD-アラニン(アラニンはアミノ酸の一種)のアミノアシル化反応の違いを自由エネルギーの観点からも議論することを可能としました。その結果、このRNAのアミノアシル化反応において、L-アラニンの反応自由エネルギーの障壁の高さは、D-アラニンに比べ9kcal/mol程低いと計算されました(図4)。このエネルギー差は実験から見積もられる値よりも大きいものの、L-アミノ酸が選択的にアミノアシル化されるという実験結果を上手く説明できます。
また、QM/MM MD計算で可視化された反応過程の分子の状態を調べたところ、遷移状態において活性部位周辺の各官能基の幾何学的な配置はL-アラニンとD-アラニンでは大きく異なっており、D-アラニンに比べL-アラニンでは遷移状態を静電的により安定化させるように各官能基が幾何学的に配置していることを見出しました(図4)。RNAミニヘリックスはtRNAへと進化したと考えられており、今回、L-アミノ酸を結合したRNAミニヘリックスが生成される反応全体のメカニズムが解明されたことで、地球上の生物は、なぜL-アミノ酸から構成されるタンパク質を使っているのかという謎の解明に大きな貢献をしました。
今後の展望
本研究は、生命の起源の謎を解明するための極めて重要な成果であり、RNAミニヘリックスにおいて、L-アミノ酸選択性を生み出すメカニズムを量子力学計算で説明できることが明らかになりました。tRNAのアミノアシル化は、現在の生物が採用しているタンパク質合成の過程において鍵となる反応であり、計算科学を用いてL-アミノ酸選択性の仕組みを解明したことによって、キラル選択的な反応を制御するタンパク質工学・核酸工学への新たな展開も期待できます。また、より高精度な量子力学計算を用いることにより、今後、より詳細で定量的なメカニズムの解明につながる可能性を秘めています。
※本研究は、日本学術振興会の科学研究費補助金(20K06592、21K06293)の助成を受けて実施したものです。
用語
*1 アミノ酸
アミノ基とカルボキシル基をもつ有機分子。タンパク質の構成単位となる。生物は通常、20種類の標準アミノ酸をもとに、タンパク質を合成する。図1参照。
*2 tRNA
運搬RNA(transfer RNA)。tRNAの一端には特定のアミノ酸が結合し、遺伝子の塩基配列に応じた適切なアミノ酸を運ぶ役割をする。RNAはヌクレオチドと呼ばれる分子が鎖状につながった構造をしており、通常、4種類の塩基を有する。
*3 アミノアシル化反応
アミノ酸を対応するtRNAに共有結合させる反応。現在の生物は、アミノアシルtRNA合成酵素がこの反応を触媒する。
*4 分子力学(Molecular Mechanics: MM)計算
古典力学に準じた単純なエネルギー関数と、量子力学計算や実験結果を再現するように最適化されたパラメータを用いて、分子の様々な構造に対して、そのポテンシャルエネルギーを計算する方法。MM計算で使用されるパラメータは力場パラメータとよばれ、1970年代から開発されているが、より高精度の計算を目指し現在でも改良が進められている。
*5 分子動力学(Molecular Dynamics: MD)法
原子・分子の運動をニュートンの運動方程式を数値的に解くことでシミュレーションする方法。ニュートンの運動方程式を解く上で必要な力の計算にはMM計算(*4)を用いることが主であり、古典力学に従った分子動力学法を、特に「古典的分子動力学法(classical MD)」と呼ぶことがある。古典的MD法は、後述する量子力学計算に比べると圧倒的に計算量が小さく、タンパク質や核酸などの生体高分子の動態を、その周りの水、細胞膜などの環境も再現しながらシミュレーションすることができる。しかしながら、古典力学に従うがゆえに、共有結合の解離・生成を伴う化学反応を扱うことができない。MD計算にはスーパーコンピュータや専用計算機を用いて、高速、かつ大規模なシミュレーションを行うことも多いが、近年はグラフィック描画に用いられるgraphics processing unit(GPU)を利用することで,比較的安価に長時間のMDシミュレーションを行うことが可能となっている。
*6 量子力学(Quantum Mechanics: QM)計算
対象の分子についてシュレディンガー方程式を数値的に解くことにより、分子のエネルギーや構造、物性などを明らかにする計算方法。QM計算は精度が高いものの、MM法に比べ圧倒的に計算量が大きく、100原子程からなる分子が計算の対象となることが一般的である。また、QM計算は電子の状態も取り扱うため、共有結合の解離・生成を伴う化学反応や、その遷移状態も扱うことが可能である。
*7 QM/MM法
化学反応が起きる重要な部分のみをQM計算で扱い、残りの大部分をMM計算で扱うことにより、計算負荷を抑えつつ、生体高分子のような巨大な分子系においても化学反応のシミュレーション解析を可能とする方法。QM/MM法を生み出したMartin Karplus、Michael Levitt、Arieh Warshelの3人は、その業績で2013年のノーベル化学賞を受賞した。
*8 アンブレラサンプリング法
反応経路に沿った反応進行度を表す抽象的な指標、座標を反応座標と呼び、その反応座標に沿った自由エネルギー変化を計算する手法。MD法と組み合わせて使われることが多い。
論文情報
雑誌名
Life
論文タイトル
Mechanism of Chiral-Selective Aminoacylation of an RNA Minihelix Explored by QM/MM Free-Energy Simulations
著者
Tadashi Ando and Koji Tamura
