ニュース&イベント NEWS & EVENTS
出芽酵母を利用した発現系でヒト由来ヒスタミンH₃受容体の活性回復に成功
~重要な創薬ターゲットであるGタンパク質共役型受容体の機構解明に貢献~
研究の要旨とポイント
- 出芽酵母はヒトの細胞と多くの共通点を持ち、ヒト由来のGタンパク質共役型受容体(GPCR)研究にも用いられてきましたが、出芽酵母を宿主として生産すると機能しないものも多いことから、手法の改善が模索されてきました。
- 今回、エラープローンPCR法(*1)を用いて、GPCRの1種であるヒト由来ヒスタミンH3受容体(H3R)の遺伝子にランダム変異を導入し、出芽酵母の細胞内で活性を持たないH3Rに活性を持たせることに成功しました。
- 本研究をさらに発展させることで、GPCRの機構解明につながり、新たな医薬品開発への貢献が期待されます。
研究の概要
東京理科大学先進工学部生命システム工学科の白石充典准教授、同大学大学院先進工学研究科生命システム工学専攻の渡邉彩水氏(2021年度修士課程修了)、中島愛海氏(2023年度修士課程1年)の研究グループは、エラープローンPCR法を用いて、ヒト由来ヒスタミンH3受容体(H3R)の遺伝子にランダム変異を導入し、出芽酵母(Saccharomyces cerevisiae)の細胞内で活性をもつ受容体を見出すことに成功しました。また、H3Rの活性に関与する変異を発見し、S. cerevisiae内で活性を持たなかったH3Rに活性を持たせることに成功しました。
Gタンパク質共役型受容体(GPCR)は、視覚、嗅覚、味覚をはじめとしたさまざまな生理機能を媒介する機能があり、多くの疾患に関与しています。そのため、重要な創薬ターゲットになっており、分子機構の解明が待たれています。しかしながら、GPCRの多くはS. cerevisiae内で発現すると、その機能が失活してしまうことが課題でした。そこで本研究グループは、GPCRの1つであるH3Rに焦点を当て、活性回復や機構解明を目的とした研究を進めてきました。
本研究では、エラープローンPCR法を用いてH3R遺伝子にランダム変異導入を行ったところ、H3Rの活性が回復した変異体を得ることができました。この変異体の解析から、4つのアミノ酸変異(L73M2.43、F193SECL2、S359Y6.36、C415R7.56)が活性の回復に寄与すると同定しました。これらの変異のうち3つは、クラスAのGPCRの活性化に関わるDRYやNPxxYモチーフ付近に位置しました。また、H3Rの活性が回復した変異体は、Gタンパク質結合の特異性やリガンド結合特性を保持していることもわかりました。
本研究成果はGPCRの作用機構の詳細を調べる、新たな医薬品や治療法の開発への貢献が期待されます。
本研究成果は、2023年9月26日に国際学術誌「Scientific Reports」にオンライン掲載されました。
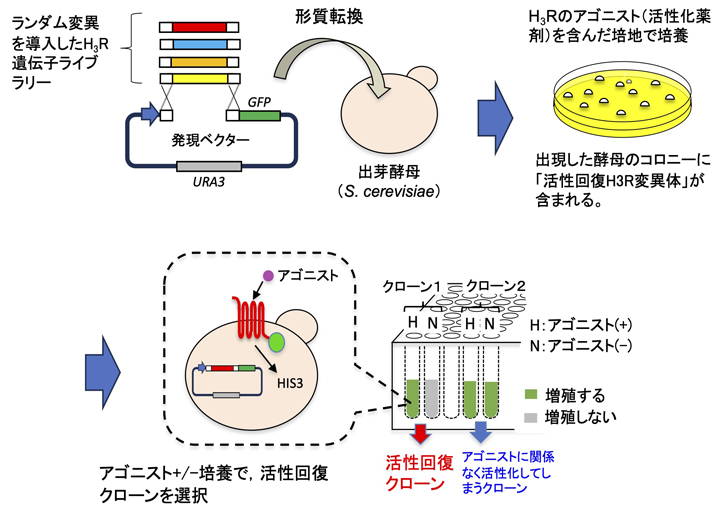
図1 本研究の概要
研究の背景
GPCRは、ヒトにおける最も多様なタンパク質群で、その数は約800種類に上ります。これらの受容体は、光、化学物質、ペプチド、タンパク質など外部からさまざまな刺激を受け取り、Gタンパク質を介して細胞にシグナル伝達を行います。視覚、嗅覚、味覚、神経伝達、免疫反応など多くの生理機能を媒介するGPCRは、さまざまな疾患とも深く関わっています。このような特性があることから、米国食品医薬品局(FDA)承認薬の30%以上がGPCRをターゲットとしています。
GPCRについては未解明のことも多く、病態解明や創薬研究を進める上で、GPCRのはたらきをより詳しく理解することは非常に重要です。そのためには、活性を持ったGPCRの構造解析や物理化学的な機能解析が必要ですが、従来報告されてきたものの多くは不活性型のGPCRに関するもので、活性型のGPCRを安定的に獲得することは困難でした。
S. cerevisiaeは、GPCRの安定変異体の選抜や機能解析のプラットフォームとして、GPCRの研究に活用されてきました。増殖が速く取り扱いが容易で、データベースやそれを利用した研究事例も豊富であることから、創薬分野を含む生命科学分野で広く活用されています。特に、ヒトGPCRの研究においては、S. cerevisiaeは宿主として使用した研究例も数多くあります。しかし、受容体との相性がありS. cerevisiae内では発現時に機能を失ってしまうものも数多くありました。
以上の背景を踏まえ、本研究ではS. cerevisiae内で発現の際に機能が消失するH3Rに焦点を当て、エラープローンPCR法でランダム変異導入を行った際の機能の変化について評価しました。
研究結果の詳細
改変型の酵母株であるYB1を、受容体のC末端にGFP(*2)を融合したH3Rで形質転換を行いました。形質転換したYB1を培養すると、GFPによる蛍光の増加が確認されました。またゲル内蛍光法によりH3Rの発現が観察されました。H3Rの形質転換体についてヒスタミンを用いたリガンド濃度依存性の増殖アッセイを行いましたが、増殖は確認されませんでした。
続いて、H3R遺伝子に対して、エラープローンPCR法によるランダム変異導入を行った後、それらの遺伝子をYB1に形質転換しヒスタミンを含む固形培地で培養したところ、複数のクローンが得られました。これらのクローンから、ヒスタミンを含む培地では増殖し、ヒスタミンを含まない培地では増殖しないクローンを得ることができました。いずれのクローンもヒスタミン濃度依存的な増殖を示したことから、7つのクローンはランダム変異導入により、機能回復したことが示唆されました。
さらに、機能回復したクローンと野生型との比較を行い、その詳細を検討しました。野生型と各クローンのGFP蛍光強度に有意差は見られず、すべてのクローンで野生型と同様に40~63 kDaの間にバンドが確認できました。ランダム変異導入とそれに続くスクリーニングの結果、4つの変異(L73M2.43、F193SECL2、S359Y6.36、C415R7.56)がシグナル伝達活性の回復に重要であることが見出されました。また、これらの変異は、GPCR活性化に重要でクラスAの GPCRの活性化に関与するDRYとNPxxYモチーフの近くに位置していることがわかりました。
最後に、H3Rに対するアゴニスト(*3)とアンタゴニスト(*4)を用いて、YB1に発現する活性が回復した変異体の機能を評価しました。その結果、野生型は活性応答を示しませんでしたが、いずれのクローンもアゴニストに対する濃度依存的な増殖を示すことが明らかとなりました。また、アンタゴニストを用いた阻害試験では、各リガンドはヒスタミン結合を阻害することにより、濃度依存的にYB1の増殖を抑制することがわかりました。特に、今回使用したアンタゴニストがインバースアゴニスト(*5)活性を示すことも明らかとなりました。
本研究では、エラープローンPCR法によるランダム変異導入と、アゴニストを含む培地での培養により、H3Rの活性を回復した変異体を得ることができました。本研究で開発された活性回復に関連する方法は、S. cerevisiaeを宿主として生産した際に活性を失う他の種のGPCRの活性回復にも応用できる可能性があり、また活性型コンフォメーションで安定化したGPCR変異体の作製にも有用であると考えられます。
本研究を主導した白石准教授は「出芽酵母(S. cerevisiae)はパンやビールなどを作るのに欠かせない微生物ですが、ヒト由来のGPCRを研究する際の宿主としても長年用いられてきました。しかし、GPCRの種類と相性があり、S. cerevisiaeの中でうまく機能しないGPCRが大半でした。このような状況を打開したいと思い、本研究に取り組んできました。今回の研究成果により、GPCRの機能解明への応用が期待できます。将来的には、副作用の少ない医薬品の開発や、現在治療法の無い疾患に対する創薬に貢献できる可能性を秘めています」と、研究の価値についてコメントしています。
※本研究は、日本学術振興会(JSPS)の科研費(JP25709080, JP15K14460)、文部科学省(MEXT)及び日本医療研究開発機構(AMED)の創薬等ライフサイエンス研究支援基盤事業(創薬等支援技術基盤プラットフォーム事業)、加藤記念バイオサイエンス振興財団の助成を受けて実施されました。
用語
*1 エラープローンPCR法
DNA増幅時に、特定の遺伝子に対してランダム変異を導入して変異体を作製する方法。
*2 GFP(Green Fluorescent Protein)
分子量約27kDaの緑色の蛍光を発するタンパク質。
*3 アゴニスト
作動薬。体内の受容体に働き、受容体を活性化する物質。
*4 アンタゴニスト
拮抗薬。体内の受容体に働き、受容体のはたらきを阻害する物質。
*5 インバースアゴニスト
アゴニストとは逆に(=inverse)、受容体を抑制するように刺激する物質。通常のアンタゴニストとは異なり、内因性の活性を持つ受容体のはたらきも抑制することが特徴。
論文情報
雑誌名
Scientific Reports
論文タイトル
Recovery of the histamine H3 receptor activity lost in yeast cells through error-prone PCR and in vivo selection
著者
Ayami Watanabe, Ami Nakajima, Mitsunori Shiroishi
