ニュース&イベント NEWS & EVENTS
単一細胞レベルで微量なRNAを高感度かつ高精度に検出、解析する新技術の開発に成功
~既存技術では検出が難しい細胞間コミュニケーションの検出も可能に~
東京理科大学
筑波大学
日本医療研究開発機構
研究の要旨とポイント
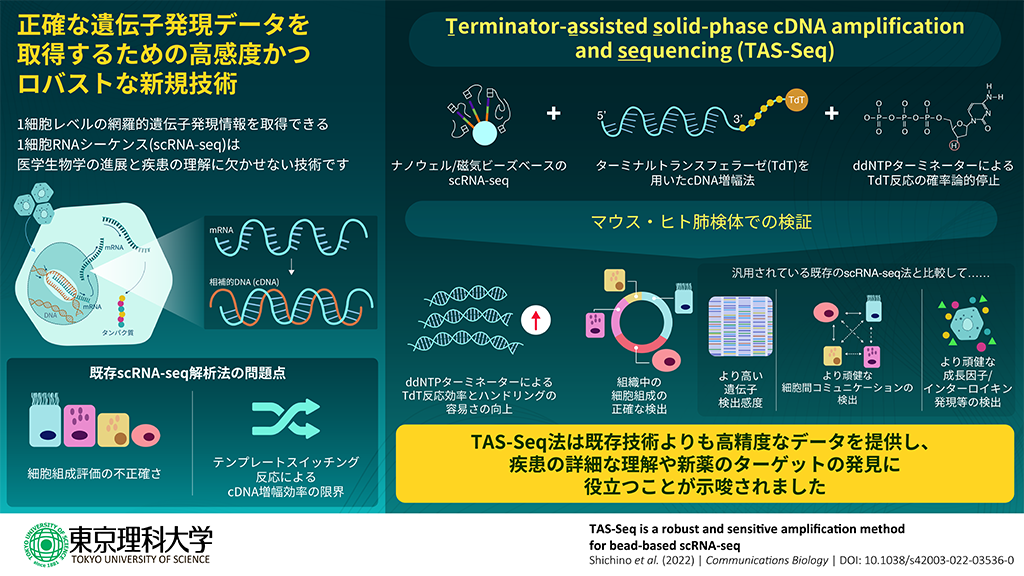
- 既存技術よりも高感度かつ高精度に1細胞中の微量なRNAを解析できる1細胞RNAシーケンス解析法「TAS-Seq法」を新たに開発しました。
- TAS-Seqは発現遺伝子の検出感度や細胞組成の定量に優れており、既存技術では検出が困難な細胞間コミュニケーションもより確実に検出することができます。
- 本研究を発展させることで、1細胞からより多くの生体内の情報を得ることができ、さまざまな疾患の発症メカニズムの解明や創薬研究の進展への貢献が期待されます。
東京理科大学生命医科学研究所炎症・免疫難病制御部門の七野成之助教、上羽悟史准教授、松島綱治教授、筑波大学プレシジョン・メディスン開発研究センターの佐藤孝明教授、和歌山県立医科大学分子病態解析研究部の橋本真一教授らの共同研究グループは、単一細胞内の微量のRNAを検出する1細胞RNAシーケンス(single-cell RNA-seq, 以下scRNA-seq)解析の新手法として、「TAS-Seq法」を開発しました。TAS-Seq法は、既存技術と比較して、発現遺伝子の検出感度や測定の頑健性、細胞組成の定量評価で非常に優れており、細胞間コミュニケーション(※1)などの生体内での重要な情報も得ることができます。
本技術は空間トランスクリプトミクス(※2)など、固相担体を用いたcDNA(※3)増幅法への応用が可能であることから、1細胞オミクス解析(※4)技術の発展、それに基づくさまざまな疾患の発症メカニズムの解明、ひいては生物学の新たな学理の構築に貢献すると期待されます。
生体におけるあらゆる細胞種とその機能を同定することが可能となるscRNA-seq解析法は、生物の仕組みを深く理解する上で必須の技術であり、近年では「ヒト細胞アトラス(※5)」計画の推進にも大きく貢献しています。この手法では、主に1細胞の遺伝子発現プロファイルの類似性に基づく細胞の分類、細胞組成に関する情報を得ることができます。scRNA-seqには既にいくつかの手法が知られていますが、遺伝子の検出感度や分析の正確性、解析系取り扱いの簡便さ(反応時間の秒単位での管理や、取り扱い中の温度上昇を厳密に避ける必要がある等)という点で課題が多く残されていました。
そこで、今回、共同研究グループは、既存技術よりも高い遺伝子検出感度に加え、反応条件許容範囲の大幅な増加による取り扱いの簡便さを兼ね備えた「TAS-Seq法」を開発しました。具体的には、ターミナルデオキシヌクレオチジルトランスフェラーゼ(TdT)(※6)を用いた高効率な固相cDNA増幅法、ターミネーターヌクレオチド(※7)による残存プライマーの伸長反応停止機構、マイクロウェル/磁気ビーズベースの細胞単離技術を組み合わせることで、新たなscRNA-seqプラットフォームを構築しました。
本研究成果を拡張することで、1細胞からのアトラス構築が可能となり、将来的には疾患解明や創薬研究の進展が期待されます。
本研究成果は、2022年6月27日に国際学術誌「Communications Biology」にオンラインで掲載されました。
研究の背景
従来のバルクシーケンスでは、複数の細胞を集団単位で分析するため、得られる情報は平均化されたものになり、各細胞の遺伝子情報や遺伝子発現のばらつきに関する情報は埋もれてしまいます。それに対して、scRNA-seqでは、1細胞レベルの遺伝子発現情報を捕捉することが可能であり、バルクシーケンスでは得られない貴重な情報を得ることができます。scRNA-seqの既存技術としては、テンプレートスイッチング法(※8)をベースとする10X Genomics社ChromiumやSmart-seq2などが知られており、広く使用されています。
しかしながら、これらの手法では、細胞組成の正確な評価、遺伝子の検出感度などにおいて、さまざまな課題がありました。例えば、マイクロ液滴技術やマイクロプレート技術を用いた細胞の単離では、サンプリングバイアス(※9)が細胞組成の結果に影響することが知られています。また、細胞に機械的な負荷がかかることによって、細胞がドロップアウト(※10)し、検出できなくなることがあります。さらに既存のTdTを用いたscRNA-seq法は高感度なものの、適切な温度・時間管理がなされない場合には、逆転写反応後の反応液中に残存するプライマー由来の副生成物が発生し、cDNA増幅法やそれ以降の分析に支障をきたすなど取り扱いに難がありました。このような課題を解決すべく、七野助教らの研究グループは、新たなscRNA-seq解析法である「TAS-Seq法」の開発に着手しました。
研究結果の詳細
本研究では、新たなscRNA-seqプラットフォームとして、「TAS-Seq法」を開発するため、以下の検討をおこないました。
1) 逆転写プライマー由来の副生成物を低減させる方法の検討
反応液中に残存するプライマー(※11)自体の増幅を抑制するため、本研究グループはターミネーター(ジデオキシヌクレオチド)に着目しました。これにより、確率論的に進むターミナルデオキシヌクレオチジルトランスフェラーゼ(TdT)のテーリング反応(※12)を抑制できるのではないかと考え、デオキシシチジン三リン酸(dCTP) (※13)、ジデオキシシチジン三リン酸(ddCTP) (※14)を添加したときのTdTのテーリング反応への影響を調べました。その結果、dCTP:ddCTP = 20 : 1にして反応させると、反応時間や反応量に関わらず、残存するプライマー由来の副生成物の長さを200bp(塩基対)未満に抑制できることがわかりました。一方で、ddCTPを添加しない場合には600bpを超えて伸長することがわかりました。TdTの反応時間を増やすと、デオキシアデニン三リン酸(dATP) (※15)のみを用いる既存のQuartz-seq2法では副生成物の長さが伸長しましたが、TAS-Seq法ではサイズ分布は変わりませんでした。以上の検討により、TAS-Seq法が既存手法よりも反応条件の許容範囲が大幅に広いことがわかりました。
2) ナノウェル/磁気ビーズベースの細胞単離システムの適用
1細胞ずつに単離するため、ナノウェル/磁気ビーズベースのBD Rhapsodyシングルセル解析システムを使用しました。この解析システムでは等張液中で、ナノウェル中への自由落下により細胞を単離するので、従来法のように、細胞に化学的、機械的負荷が掛からず、繊細な細胞にも適用できることが示唆されました。
次に、これらの検討を踏まえて開発したTAS-Seq法を用いて、マウス肺、脾臓、腎臓組織などのさまざまな細胞での解析データを取得し、10X Chromium v2やSmart-seq2などの既存の手法で得られたデータと比較して、その特性を評価しました。
その結果、TAS-Seq法は、既存の手法に比べて、シーケンスの量の多寡に関わらず、より多くの遺伝子や変動性の高い遺伝子(highly-variable genes)を高感度に検出できることが示されました。
次にマウスの肺組織を使って、細胞組成の定量の観点からの評価を行ったところ、CD4+, CD8+T細胞サブセットについて、TAS-Seq法は既存の手法よりも良い分離を示しました。また、既存の手法では細胞の割合が異なる、検出できない細胞があるなどの問題が生じましたが、TAS-Seq法では細胞を正確に捕捉できることがわかりました。同様に、ヒト間質性肺疾患患者の肺組織でも、TAS-Seq法では正確な細胞組成を得られました。さらに、遺伝子のドロップアウト率も、既存の手法と比較して低いことがわかりました。TAS-Seq法は発現が不十分な遺伝子に対する感度が高いため、mRNA含有量量の低い細胞(例えば好中球や好塩基球)を効率的に検出することができると考えられます。
最後に、マウス肺のデータを用い、細胞間コミュニケーションの分析を行い、既存の手法との違いを検討しました。scRNA-seq解析データでは、個々の細胞が発現している遺伝子を捉えることができるので、ある細胞Aと細胞Bが、細胞外で相互作用することが知られている分子Xとその受容体Yを発現しているかの情報が得られます。そのようなありうる相互作用を全て検索し統計解析することで、検体中で統計的に意味のある細胞間コミュニケーションを網羅的にとらえ、また細胞同士の相互作用の強さも推定することができます。TAS-Seq法では既存の手法と比較して、細胞間コミュニケーションを最も多く捉えられることがわかりました。実際、TAS-Seq法でのみ、肺の恒常性、修復、および再生に重要な役割を果たしていることが知られている、2型肺胞上皮細胞、肺胞線維芽細胞、血管内皮細胞間の強い相互作用を同定することができました。
本研究で開発されたTAS-Seq法を用いた解析により、既存技術と比較して高精度なデータを取得することができ、重要な細胞間コミュニケーションも捉えられるようになることから、新たな疾患の治療標的の発見に役立つ可能性があります。また本技術は、組織切片上での網羅的遺伝子発現解析技術である空間トランスクリプトミクスにも応用可能であり、今後益々の発展が予想される1細胞オミクス技術の発展、それに基づく疾患原理や生物学の理解の促進に大きく貢献すると期待されます。
※本研究は日本医療研究開発機構(AMED)の革新的先端研究開発支援事業(PRIME)「生体組織の適応・修復機構の時空間的解析による生命現象の理解と医療技術シーズの創出」研究開発領域における研究開発課題「細胞間相互作用テンポラルネットワーク解析による線維化肺修復機構の解明」(研究開発代表者:七野成之)(JP21gm6210025)、日本学術振興会(JSPS)科研費の新学術領域研究(17H06392)、若手研究(19K16620)の助成を受けて実施されました。
※本論文で発表した「TAS-Seq法」をベースに、本研究グループは既にその反応系の各ステップを最適化・改良した「TAS-Seq2法」の開発に成功しました。TAS-Seq2法では、マウス脾臓細胞でTAS-Seq法と比べ、約1.5~2倍の遺伝子検出感度の向上を達成しています。本研究成果の社会還元の一環として、本技術をもとに東京理科大学発ベンチャー企業イムノジェネテクス社を2019年に設立し、「TAS-Seq/TAS-Seq2法」を用いたscRNA-seq解析サービスを提供しており、すでに安定稼働を実現しています。
東京理科大学発ベンチャー企業イムノジェネテクス社
URL:http://immunogeneteqs.com/
用語
※1 細胞間コミュニケーション:多細胞生物が恒常性を保つため、細胞間で行われる情報伝達。サイトカインやケモカイン、成長因子の分泌や、細胞間接着、神経伝達を介するものなど様々な伝達方法が知られている。
※2 空間トランスクリプトミクス:空間的遺伝子発現解析。組織切片をmRNAキャプチャー用プライマーがスポットされたスライドグラス等に貼り付け、スライド上で組織溶解・mRNA補足・cDNA合成を行うことで、組織中における位置情報を保持した網羅的遺伝子発現情報を得ることができる技術。スライドグラス等のスポットごとに異なるDNAバーコードがあるので、それらDNAバーコードを用いて照合することにより、組織上のスポット単位の遺伝子発現が得られる。このデータを分析することで、遺伝子発現の空間的分布を網羅的に得られる。
※3 cDNA(相補的DNA):RNAから逆転写反応で合成されたDNA。
※4 オミクス解析:生体内の分子を網羅的に調べること。対象が1細胞の場合を、「1細胞オミクス解析」という。
※5 ヒト細胞アトラス:人体を構成する細胞全ての分類やマッピングを行う国際共同プロジェクト。
※6 ターミナルデオキシヌクレオチジルトランスフェラーゼ(TdT):鋳型に依存せずに、DNAの3'末端にデオキシリボースを持ったヌクレオチドを連結させる酵素。
※7 ターミネーターヌクレオチド:DNA合成反応を停止させる役割をもつヌクレオチド。
※8 テンプレートスイッチング法:鋳型(テンプレート)となるRNAの5'末端の配列が未知(あるいは共通の配列を有していない)の場合でも、そのRNAを鋳型としてRT-PCR増幅を可能とする技術。
※9 サンプリングバイアス:サンプリングに偏りが生じることにより、真値から外れた結果が得られること。
※10 ドロップアウト:目的の細胞や遺伝子が消失すること。
※11 プライマー:DNAを増やすために用いる短い人工的なDNA
※12 テーリング反応:cDNAの3'末端に人工的にDNA配列を追加する反応。細胞から合成したcDNAの3'末端配列は極めて多様性に富んでいるため、人工的に決まったDNA配列を付加することで、プライマーを用いたPCR反応によるcDNA増幅が可能となります。
※13 デオキシシチジン三リン酸(dCTP):DNAを合成する際の素材となる、DNAを構成する主な4つの塩基(アデニン(A),チミン(T), グアニン(G), シトシン(C))のうちの一つ。
※14 ジデオキシシチジン三リン酸(ddCTP):dCTPは3'末端に-OH基がついており、DNA合成中に取り込まれた際に、-OH基を介して次の塩基へとつながって伸長することができる。一方ddCTPはdCTPの3'末端の-OH基が欠損した物質であり、一度取り込まれると、それ以上のdCTPなどの塩基の伸長反応が起こらなくなり、DNAの伸長が停止することになる。
※15 デオキシアデニン三リン酸(dATP):DNAを合成する際の素材となる、DNAを構成する主な4つの塩基(アデニン(A),チミン(T), グアニン(G), シトシン(C))のうちの一つ。
論文情報
雑誌名
Communications Biology
論文タイトル
TAS-Seq is a robust and sensitive amplification method for bead-based scRNA-seq
著者
Shigeyuki Shichino, Satoshi Ueha, Shinichi Hashimoto, Tatsuro Ogawa, Hiroyasu Aoki, Bin Wu, Chang-Yu Chen, Masahiro Kitabatake, Noriko Ouji-Sageshima, Noriyoshi Sawabata, Takeshi Kawaguchi, Toshitugu Okayama, Eiji Sugihara, Shigeto Hontsu, Toshihiro Ito, Yasunori Iwata, Takashi Wada, Kazuho Ikeo, Taka-aki Sato, and Kouji Matsushima
DOI
発表者
七野 成之 東京理科大学 生命医科学研究所 炎症・免疫難病制御部門 助教
上羽 悟史 東京理科大学 生命医科学研究所 炎症・免疫難病制御部門 准教授
松島 綱治 東京理科大学 生命医科学研究所 炎症・免疫難病制御部門 教授
佐藤 孝明 筑波大学 プレシジョン・メディスン開発研究センター センター長
橋本 真一 和歌山県立医科大学 分子病態解析研究部 教授
お問い合わせ
【研究に関する問い合わせ先】
東京理科大学 生命医科学研究所 炎症・免疫難病制御部門 助教
七野 成之(しちの しげゆき)
E-mail:s_shichino【@】rs.tus.ac.jp
【報道・広報に関する問い合わせ先】
東京理科大学 広報部 広報課
TEL:03-5228-8107 FAX:03-3260-5823
E-mail:koho【@】admin.tus.ac.jp
筑波大学 広報局
TEL:029-853-2040 FAX:029-853-2014
E-mail:kohositu【@】un.tsukuba.ac.jp
【AMED事業に関する問い合わせ先】
日本医療研究開発機構(AMED)
シーズ開発・研究基盤事業部 革新的先端研究開発課
TEL:03-6870-2224
E-mail:kenkyuk-ask【@】amed.go.jp
【@】は@にご変更ください。
研究室
松島研究室のページ:https://k-matsushimalab.org/
松島教授のページ:https://www.tus.ac.jp/academics/teacher/p/index.php?6FA1
上羽准教授のページ:https://www.tus.ac.jp/academics/teacher/p/index.php?7037
七野助教のページ:https://www.tus.ac.jp/academics/teacher/p/index.php?7039
東京理科大学について
東京理科大学:https://www.tus.ac.jp/
詳しくはこちら
